Allgemeine Kontaktinformationen
Anschrift:
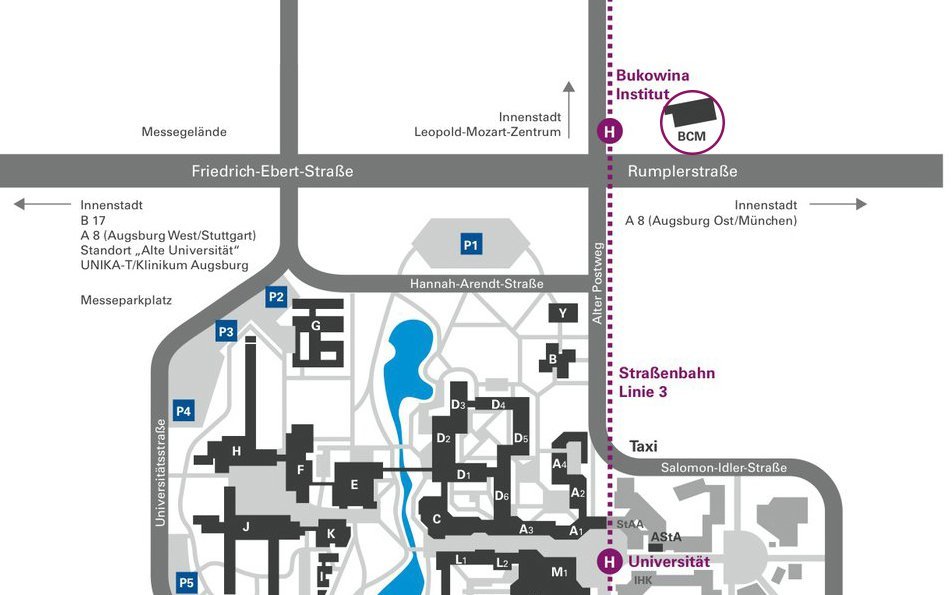
Lehrstuhl für Translationale Medizinische Forschung
Fakultät für Angewandte Informatik
Universität Augsburg
Alter Postweg 101
86159 Augsburg
Sekretariat:
Telefon: +49 821 598 -3738
Fax: +49 821 598 -3739
E-Mail: sekretariat-misit@informatik.uni-augsburg.de
Gebäude:
Büro Center Messe (BCM) - 9. Stock